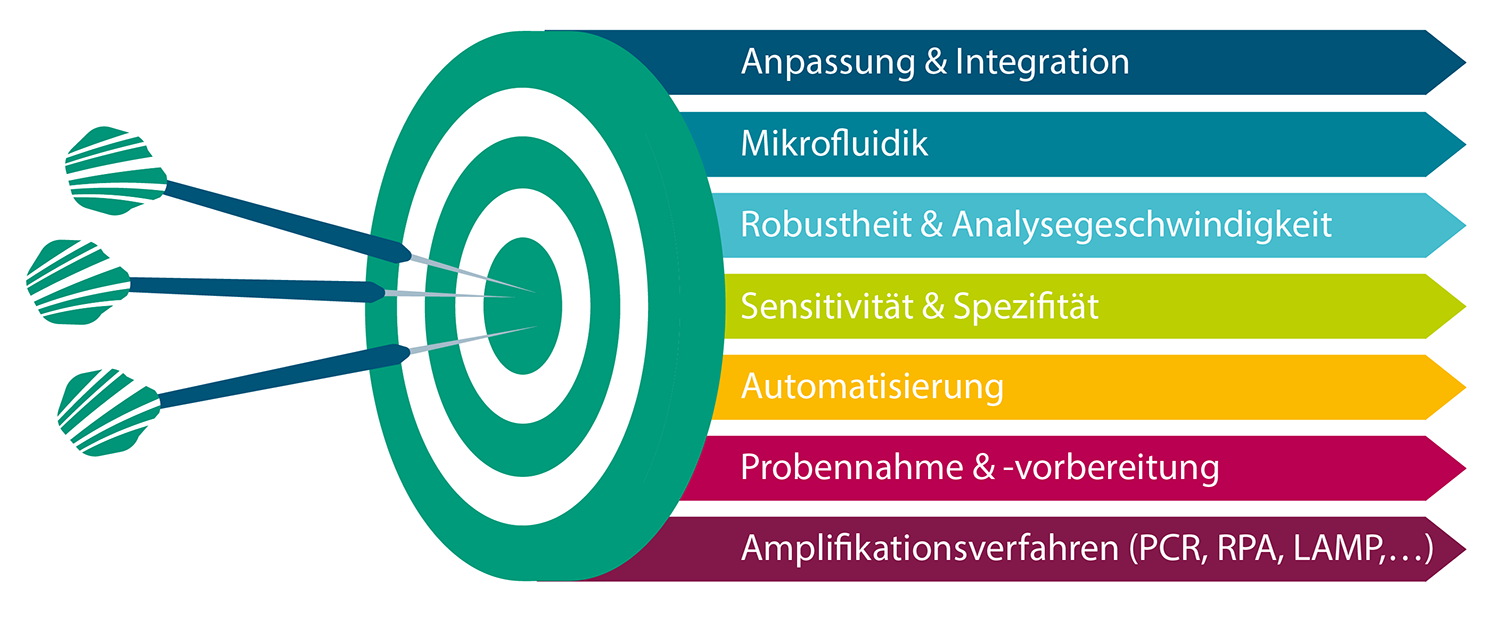
- Sicherheit und Flexibilität: Die Integration von Mikrofluidik in automatisierten Systemen erlaubt eine einfache, benutzerfreundliche und flexible Handhabung der Technologie.
- Verlässliche Analysen: Mikrofluidik-basierte Technologien bieten eine präzise Kontrolle über die Reaktionsbedingungen, was zu einer hohen Genauigkeit und Reproduzierbarkeit der Ergebnisse führt.
- Zeit- und Kosteneffizienz: Automatisierte Mikrofluidik-basierte Technologien ermöglichen schnelle, Material-sparsame Amplifikationsprozesse und minimieren den Arbeitsaufwand.
Lab-on-chip PCR und isotherme Amplifikation
Häufige Fragen:
Welche Nukleinsäureamplifikationsverfahren lassen sich mikrofluidisch umsetzen?
Für den Transfer vom konventionellen in ein mikrofluidisches System kommen grundsätzlich alle gängigen Amplifikationsverfahren in Betracht. Aktuelle Projekte am Fraunhofer IMM beschäftigen sich schwerpunktmäßig mit der RT-qPCR und qRPA on-chip, in sowohl single-, als auch multiplex-Ansätzen.
Welche Targets lassen sich nachweisen? Welche Proben können untersucht werden?
Mit Hilfe von Mikrofluidik-basierten Nukleinsäureamplifikationstechnologien lassen sich frei wählbare Abschnitte eines beliebigen DNA-Strangs hochspezifisch vervielfältigen und detektieren. Dabei können grundsätzlich sämtliche Proben, die Zellen von Tieren, Pflanzen, Pilzen oder Mikroorganismen, oder zellfreie Nukleinsäure enthalten, zum Einsatz kommen. Ein Forschungsschwerpunkt am IMM umfasst Strategien zur Reduktion, Vereinfachung und Au-tomatisierung erforderlicher Probenvorbereitungsschritte.
Welche Anwendungsbeispiele wurden bisher untersucht? Welchen Reifegrad haben bisher entwickelte Systeme?
Am Fh IMM wurde ein Plattform-Prototyp für die automatisierte RT-PCR angepasst an den SARS-CoV-2 Nachweis entwickelt. In Zusammenarbeit mit dem Institut für Virologie der Uni-versitätsmedizin Mainz wurden mehrere 100 Patientenproben im Hinblick auf die diagnosti-sche Leistungsfähigkeit untersucht. Das System besitzt TRL 6 an der Schwelle zur Produktentwicklung. In einem aktuellen und vom Bundesministerium für Bildung und Forschung geförderten Ver-bundprojekt forschen Fh Wissenschaftler*innen gemeinsam mit Partnern aus Akademie und Wirtschaft an einem System zur quantitativen isothermen "on-chip"-Amplifikation zellfreier DNA. Dieses Vorhaben umfasst unter anderem eine frühe klinische Studie in Zusammenarbeit mit der Klinik für Anästhesiologie der Universitätsmedizin Mainz.
 Fraunhofer-Institut für Mikrotechnik und Mikrosysteme IMM
Fraunhofer-Institut für Mikrotechnik und Mikrosysteme IMM